Cette étude est faite à partir de données moléculaires concernant la cytochrome c oxydase.
Des données semblables sont disponibles sur le site phylogène de l’INRP.
Télécharger le fichier

cytocoxydase.pir (clic droit sur le lien) , si celui-ci s’enregistre sur le disque avec la double extension .pir.txt supprimer cette dernière dans l’explorateur de Windows. Placer ce fichier dans le dossier molécules puis primates du logiciel phylogène.
Dans phylogène cliquer sur Fichier, Ouvrir, Tableau de molécules, sélectionner dans la liste déroulante Molécules non alignées (PIR) et ouvrir le dossier primates. Sélectionner le fichier cytocoxydase.pir.
Cliquer sur Aligner, les molécules sont comparées à la séquence de référence qui est en haut, les similitudes sont en rouge, les différences en noir.
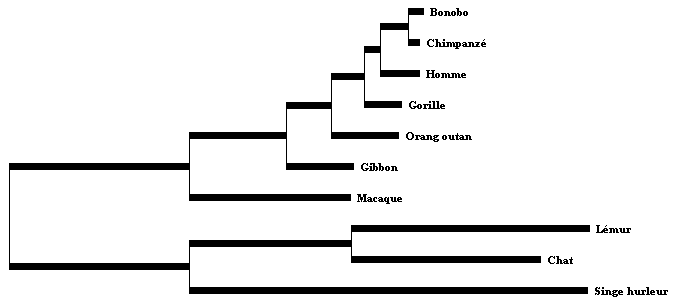
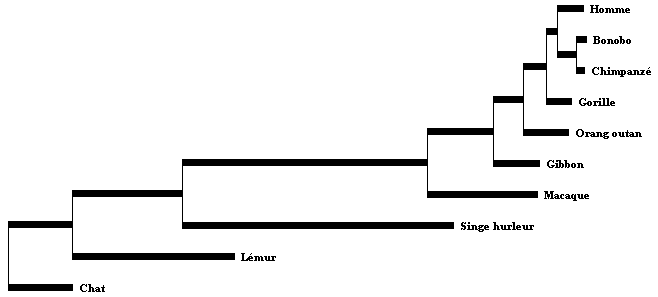
Cliquer sur Arbre. Par défaut l’arbre est tracé en UPGMA qui ne permet pas de l’enraciner. Cliquer sur Options et choisir l’option NJ. Toutes les espèces appartiennent aux primates à l’exception du chat qui est l’espèce extérieure sur laquelle l’arbre sera enraciné.
Arbre non enraciné :
Arbre enraciné sur l’espèce chat :
Passer la souris sur les branches de l’arbre pour voir apparaître les noms des groupes (inutile de cliquer, il n’y a pas de lien).
Des molécules appartenant à d’autres espèces sont disponibles sur le site NCBI, il faut choisir la rubrique "Protein" et demander la recherche de cytochrome c oxidase polypeptide II suivi, éventuellement, du nom de l’espèce en anglais ou en latin.
Un autre arbre peut être construit en utilisant les séquences du gène du récepteur au PTC (PhénylThioCarbamide) :


