Proposition d’un fichier de séquences permettant la construction d’un arbre phylogénétique avec Phylogène ou Geniegen2 pour montrer un transfert horizontal entre bactéries et papillons.
Auteur : Vincent GUILI, Lycée Descartes, Saint-Genis-Laval
Liens avec le BO
BO spécial n°8 du 25 juillet 2019
Terminale spécialité : La Terre, la vie et l’organisation du vivant
Génétique et évolution
La complexification ds génomes : transferts horizontaux et endosymbioses
L’universalité de l’ADN et l’unicité de sa structure dans le monde vivant autorisent des échanges génétiques entre organismes non nécessairement apparentés.
Des échanges de matériel génétique, hors de la reproduction sexuée, constituent des transferts horizontaux. Ils se font par des processus variés (vecteurs viraux, conjugaison bactérienne...).
Les transferts horizontaux sont très fréquents et ont des effets très importants sur l’évolution des populations et des écosystèmes. Les pratiques de santé humaine sont concernées (propagation des résistances aux antibiotiques).
Mise en situation
De nombreuses plantes produisent des produits chimiques appelés glycosides cyanogènes qui peuvent être convertis en cyanure lorsque la plante est mangée. C’est une défense ancienne et efficace contre toutes sortes d’herbivores. Cependant certains insectes et acariens sont capables de se développer sur les plantes cyanogènes. Le gène Tu-CAS permet la synthèse d’une enzyme qui intervient dans le métabolisme de la cystéine, mais aussi dans la détoxification du cyanure. Il est présent chez les plantes et chez les bactéries, mais a aussi été identifié chez des arthropodes résistants au cyanure.
En comparant les séquences de ce gène, il est possible de reconstruire un arbre phylogénétique, chaque branche correspondant à une espèce.
Vue d’ensemble schématique de la voie de biosynthèse de la cystéine
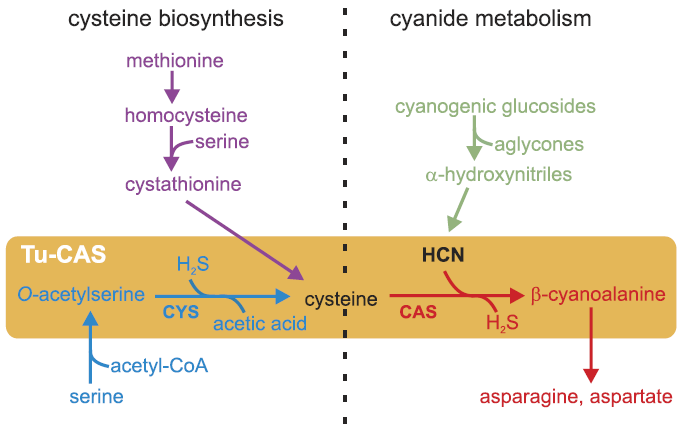
Source : https://doi.org/10.7554/eLife.02365
Vue d’ensemble schématique de la voie de biosynthèse de la cystéine chez les Métazoaires (en violet) et les Plantes/Bactéries (en bleu), de la libération de HCN pendant la cyanogénèse des plantes (en vert) et de la principale voie de détoxication de HCN chez les arthropodes (en rouge).
Les deux réactions catalysées par le produit du gène Tu-CAS sont marquées par un fond orange et sont indiquées par CYS et CAS. Le CAS détoxifie le cyanure par incorporation dans la cystéine, formant ainsi la β-cyanoalanine, qui peut être métabolisée davantage par les nitrilases. La CYS catalyse la deuxième étape de la voie de synthèse de la cystéine dans les bactéries et les plantes, après que la sérine soit convertie en O-acétylsérine.
Référence
Wybouw, N., Dermauw, W., Tirry, L., Stevens, C., Grbić, M., Feyereisen, R., & Van Leeuwen, T. (2014). A gene horizontally transferred from bacteria protects arthropods from host plant cyanide poisoning. eLife, 3, e02365. https://doi.org/10.7554/eLife.02365
Fichier de séquences : Tu-CAS.edi
Ce fichier contient les séquences protéiques correspondant aux gènes de Cystéine Synthétase des espèces suivantes :
- Soja
- Pomme de Terre
- Maïs
- Bactérie Achromobacter xylosoxidans
- Bactérie Clostridium difficile
- Bactérie Mesorhizobium opportunistum
- Bactérie Methylobacterium radiotolerans
- Bactérie Thermovirga lienii
- Bactérie Sagittula stellate
- Bombyx du murier
- Noctuelle verdoyante
- Papilio xuthus
- Champignon Aspergillus fumigatus
La séquence du champignon sert d’extra-groupe, ce qui permet d’enraciner l’arbre.
Les séquences représentent un échantillon de celles utilisées dans la publication de référence.
Téléchargement : Tu-CAS.edi (clic-droit, Enregistrer la cible du lien sous...)
Procédure pour construire l’arbre phylogénétique avec Phylogène
- lancer Phylogène
- ouvrir les séquences : Fichier/Ouvrir/Fichier de molécules
- dans la nouvelle fenêtre qui s’ouvre : Fichier/Ouvrir et choisir Types de Fichiers : Fichier Anagène (EDI)
- sélectionner toutes les séquences (bouton en bas de fenêtre)
- aligner les séquences (bouton à droite)
- possibilité de colorer les séquences : barre de menu couleur/Colorer les séquences
- sélectionner le type d’arbre : bouton Options (à droite) et choisir Arbre UGPMA ou NJ
- afficher l’arbre : bouton Arbre à droite
- possibilité de rotation d’une branche autour de son axe avec l’outil

(ne change pas les relations phylogénétiques). Cela permet de visualiser plus clairement la position des gènes des papillons au milieu de ceux des bactéries.
Résultats
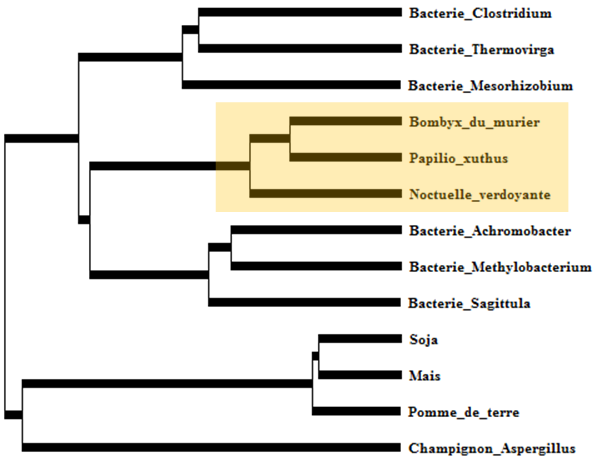
Arbre UGPMA
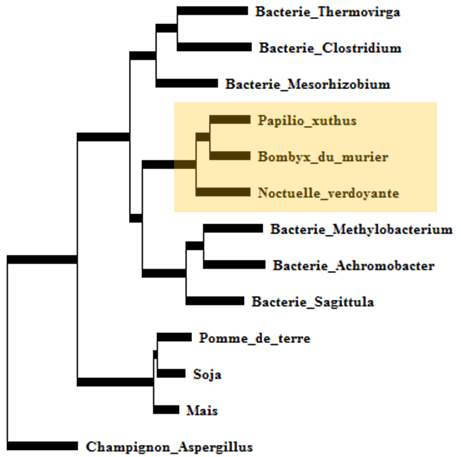
Arbre NJ
Remarque sur les méthodes de construction d’arbre
La méthode UPGMA (Unweight Pair Group Method with Arithmetic mean) est utilisée pour reconstruire des arbres phylogénétiques si les séquences ne sont pas trop divergentes. Il y a d’abord identification des deux séquences les plus proches et ce groupe est ensuite traité comme un tout, puis on recherche la séquence la plus proche et ainsi de suite jusqu’à ce qu’il n’y ait plus que deux groupes.
La méthode NJ (Neighbor-Joining) tente de corriger la méthode UPGMA afin d’autoriser un taux de mutation différent sur les branches. Les données initiales permettent de construire une matrice qui donne un arbre en étoile. Cette matrice de distances est ensuite corrigée afin de prendre en compte la divergence moyenne de chacune des séquences avec les autres.
L’arbre est alors reconstruit en reliant les séquences les plus proches dans cette nouvelle matrice. Lorsque deux séquences sont liées, le nœud représentant leur ancêtre commun est ajouté à l’arbre tandis que les deux feuilles sont enlevées. Ce processus convertit l’ancêtre commun en un nœud terminal dans un arbre de taille réduite.
Voir : http://www.info.univ-angers.fr/ gh/Idas/Wphylog/infobiogen/phylogenie.htm
Procédure pour construire l’arbre phylogénétique avec Geniegen 2
- ouvrir Geniegen 2
- charger les séquences : Séquences/Charger des séquences
- sélectionner toutes les séquences
- aligner les séquences : Actions/Aligner les séquences sélectionnées
- afficher l’arbre : Affichage/Afficher le phénogramme
Alternativement, en cliquant sur ce lien, on ouvre directement les séquences dans Geniegen2, et il n’y a plus qu’à aligner et afficher l’arbre.
Résultats
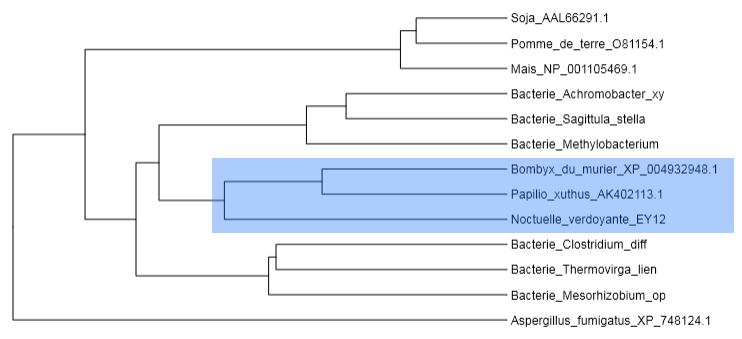
Phénogramme obtenu à partir des séquences de l’enzyme Tu-CAS
Le gène Tu-CAS est normalement absent chez les animaux. Celui présent chez les papillons est apparenté à celui des bactéries : ce gène a été transféré des bactéries aux papillons. Il permet aux chenilles de se nourrir sur des plantes qui produisent du cyanure d’hydrogène.
NB : Certaines espèces de papillons, comme Heliconius melpomene ou Danaus plexippus se sont mêmes mises à produire elles-mêmes du cyanure d’hydrogène. La cyanogenèse sert à la défense contre les prédateurs, mais stocke également de l’azote réduit qui peut être mobilisé pour la synthèse de la chitine, et joue un rôle dans le choix du partenaire en déterminant l’attrait des cadeaux nuptiaux du mâle vers la femelle.
Les séquences les plus proches des Tu-CAS des papillons sont celles d’α- et β-protéo-bactéries comme les Methylobacterium. Ces bactéries vivent à la surface des plantes mais établissent aussi des colonies endophytes. Lors de la consommation de la plante par un insecte phytophage, elles peuvent survivre à l’intérieur des arthropodes hôtes. Par ailleurs, les protéo-bactéries sont aussi des endosymbiotes connus des arthropodes et résident souvent dans les organes reproducteurs, ce qui leur assure une transmission verticale aux générations suivantes. Ces deux éléments sont susceptibles de faciliter un transfert horizontal de gènes.
Complément
Proposé par Hervé FURSTOSS, Lycée Louis Armand, Mulhouse
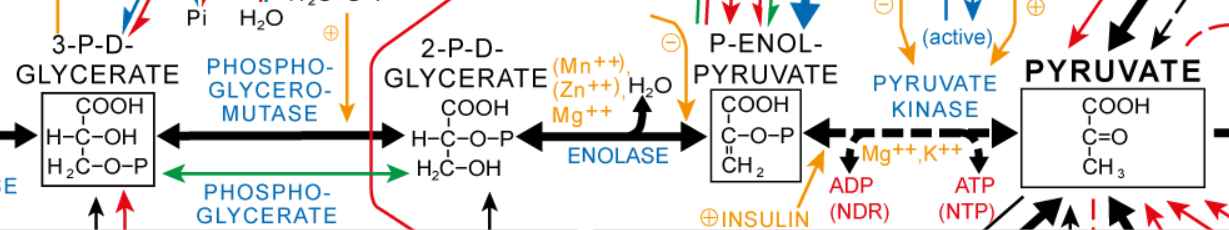
Pour montrer l’incongruité de l’arbre obtenu par rapport à la phylogénie des différents groupes d’êtres vivants, on peut le comparer à un phénogramme construit à partir d’un gène n’ayant pas fait l’objet de transfert horizontal. Le gène de l’énolase est un bon candidat car il est quasi-ubiquiste, donc d’origine ancienne, et les séquences sont connues pour de nombreux organismes. L’énolase est une enzyme qui catalyse une étape de la glycolyse :
Détail de la glycolyse
L’énolase (au centre) catalyse l’avant dernière réaction, permettant la conversion du 2-phospho-D-glycérate (2PG) en phosphoénolpyruvate (PEP).
Source : Biochemical Pathways, Roche
Le fichier sequences_enolase.edi (clic-droit, Enregistrer la cible du lien sous...) contient les séquences protéiques de l’énolase pour la plupart des organismes présents sur le phénogramme de Tu-CAS sauf la pomme de terre (remplacée par la tomate) et la Noctuelle verdoyante non disponible sur Genbank. Il y a 3 organismes supplémentaires dans le fichier : la drosophile et l’être humain pour relier les papillons à d’autres animaux connus des élèves, et le tétranyque (acarien parasite des végétaux qui a donné son nom à l’enzyme Tu-CAS). Dans les commentaires du fichier figurent les noms d’espèce et la référence Genbank des différentes séquences.
Après alignement des séquences avec Geniegen2 (lien direct pour afficher les séquences), on peut construire l’arbre suivant :
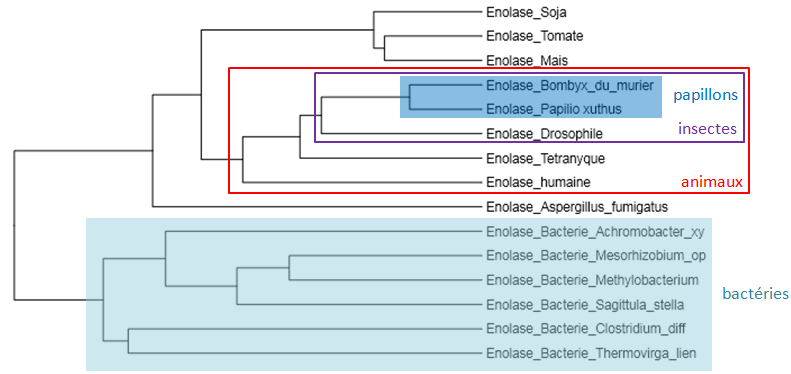
Phénogramme obtenu à partir des séquences de l’énolase
L’arbre obtenu correspond à des transferts verticaux du gène de l’énolase génération après génération, et donne les relations phylogénétiques entre les différentes espèces. La comparaison avec l’arbre obtenu avec Tu-CAS permet de bien montrer le transfert horizontal.