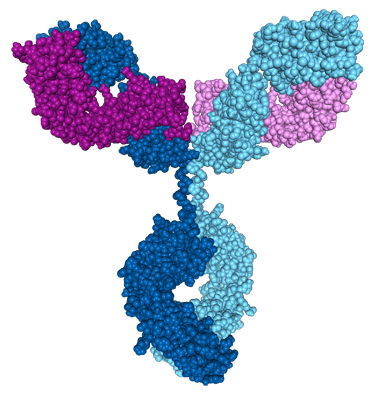
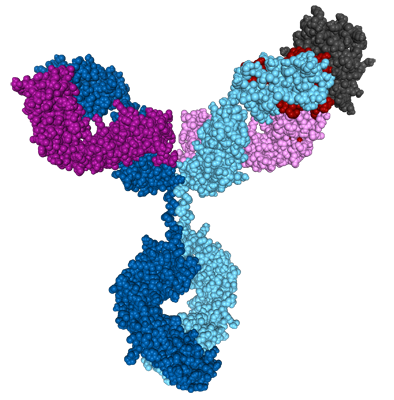
Cette page présente la structure complète d’un anticorps en complexe avec un antigène, et non pas seulement un fragment comme c’est habituellement le cas.
Observation d’une molécule d’immunoglobuline
Fichier de structure à télécharger :

L’affichage des différentes chaines dans Libmol met en évidence les deux chaînes lourdes et les deux chaînes légères. Lien direct en cliquant sur l’image.
Comparaison des chaînes d’une immunoglobuline
Les séquences protéiques proposées peuvent être comparées avec Anagène ou GénieGen.
Fichier à télécharger (Clic droit : Enregistrer la cible du lien sous... ) :

igg.edi : contient les séquences des quatre chaînes d’une même immunoglobuline.
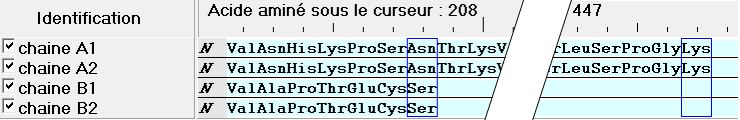
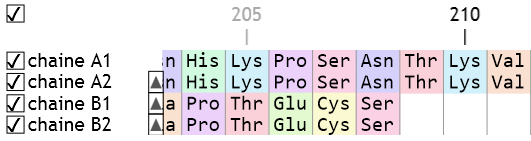
Une comparaison simple des séquences met en évidence la présence de 2 chaines courtes B1 et B2 identiques et de 2 chaines longues A1 et A2 identiques. Les chaines courtes et longues étant par ailleurs différentes.
Cette comparaison peut être réalisée en ligne avec Geniegen 2, sans nécessité de télécharger les séquences puisqu’elles sont incluses dans la banque. Lien direct en cliquant sur l’image :
Mise en évidence des zones hypervariables
Fichier à télécharger (Clic droit : Enregistrer la cible du lien sous... ) :

chaine_h.edi contient les séquences de 8 chaînes lourdes d’individus différents.
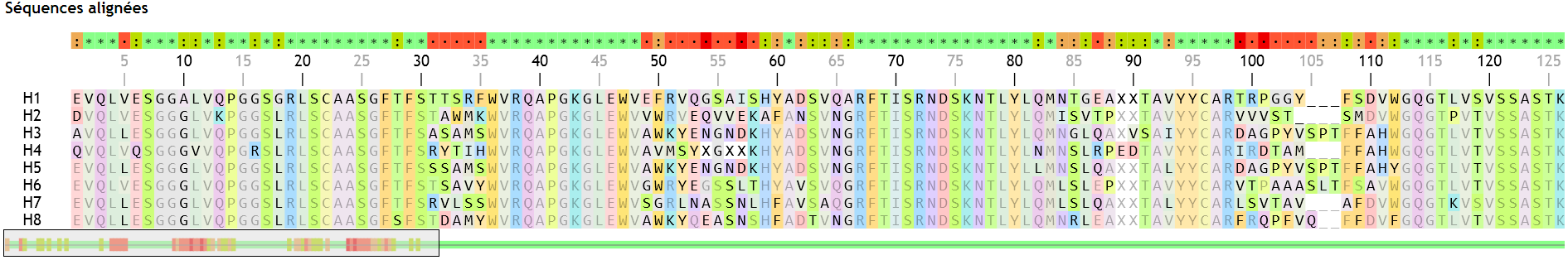
Une comparaison de toutes les chaînes lourdes montre des zones hypervariables localisées dans la première partie des protéines, alors que le reste est constant :
L’alignement est obtenu en ligne avec GenieGen2. Lien direct en cliquant sur l’image.
Les positions où l’on trouve au moins 4 acides aminés différents pour les 8 chaînes sont les suivantes (au survol par la souris, Geniegen2 indique la position originale d’un acide aminé avant alignement) :
1, 31-35, 49-58, 85, 87, 90, 99-104
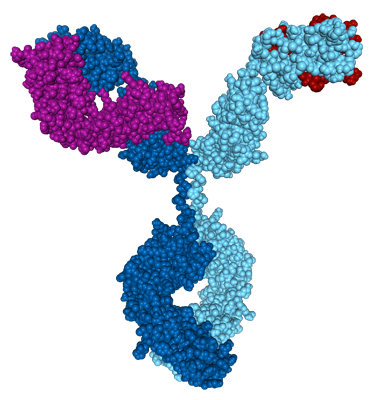
Ces acides aminés hypervariables peuvent être mis en évidence (ici en rouge) sur la structure de l’immunoglobuline :
Fichier à télécharger (Clic droit : Enregistrer la cible du lien sous... ) :

chaine_l.edi contient les séquences de 9 chaînes légères d’individus différents.
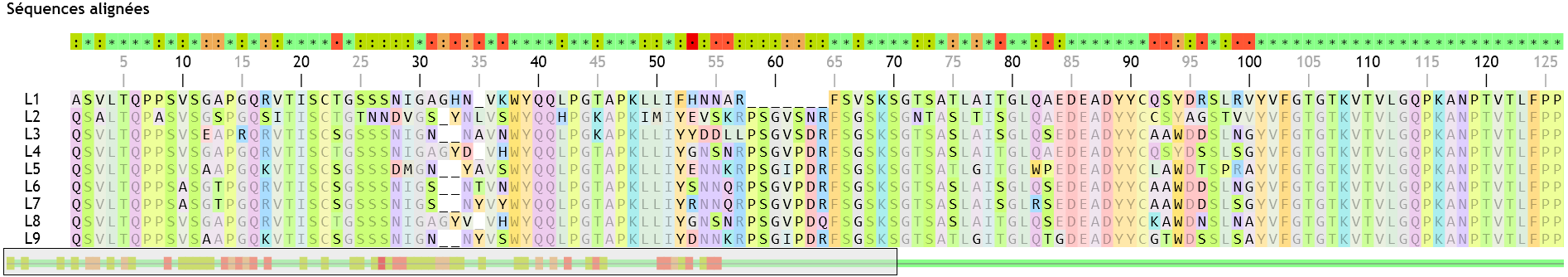
Une comparaison de toutes les chaînes légères montre là aussi des zones hypervariables localisées dans la première partie des protéines, alors que le reste est constant :
L’alignement est obtenu en ligne avec GenieGen2. Lien direct en cliquant sur l’image.
Les positions où l’on trouve au moins 4 acides aminés différents pour les 9 chaînes sont les suivantes (au survol par la souris, Geniegen2 indique la position originale d’un acide aminé avant alignement) :
33-34, 36, 52, 81, 90, 94, 98
Ces acides aminés hypervariables peuvent là encore être mis en évidence (en rouge) sur la structure de l’immunoglobuline :
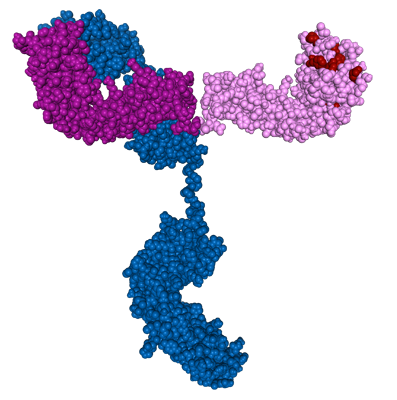
Fixation de l’antigène
Fichiers de structure à télécharger :

Complexe_Anticorps_Antigene.pdb est la structure d’une IgG en complexe avec un antigène, la nucléase du staphylocoque (modèle 1NSN).
Il est important de noter que ce modèle de structure dans son ensemble n’a pas été obtenu à partir d’un complexe immun cristallisé. En effet les deux bras d’un anticorps sont très flexibles à leur jonction avec la partie Fc (les deux chaines lourdes du bas). Cela rend très difficile la cristallisation d’anticorps intacts. C’est pour cette raison que les structures de complexes anticorps-antigène présentes dans les bases de données PDB ne contiennent que des fragments des bras supérieurs (Fv, Fab ou F(ab)’2). Le modèle proposé a été construit avec le logiciel Hex Protein Docking. L’ajustement des domaines variables (partie Fv) sur la partie constante de l’anticorps a été réalisé de façon empirique, "à la main", créant ainsi une structure chimérique uniquement pour des visées didactiques. Il ne s’agit donc pas d’un modèle complètement issu de travaux de recherche.
L’antigène peut être affiché (ici en noir) dans Libmol :
Les zones hypervariables des chaines de l’immunoglobuline sont au contact de l’antigène, ce qui permet de relier la spécificité de l’anticorps à sa structure. Lien direct en cliquant sur l’image.
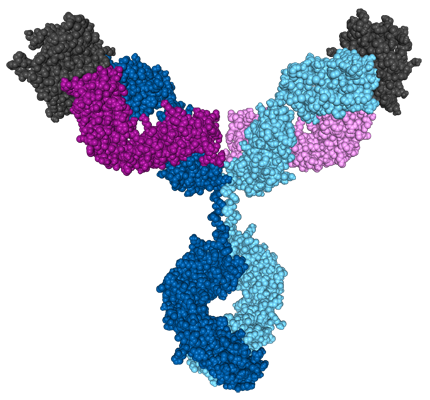
L’affichage de toutes les chaines dans Libmol met en évidence 2 antigènes (en noir) au contact de l’immunoglobuline :
Cette immunoglobuline est divalente : elle possède deux sites identiques de liaison à l’antigène, un au bout de chaque bras. Elle peut donc se lier à deux molécules d’antigène. Lien direct en cliquant sur l’image.
Auteurs : Jacques JANIN et Vincent GUILI