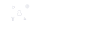Étude de la myoglobine
La myoglobine est une protéine qui fixe le dioxygène dans les muscles.
On peut rechercher les liens de parenté entre des vertébrés en utilisant les séquences de cette molécule. Ouvert dans Phylogène, le fichier myoglobine.pir, permet, après alignement, d’observer la matrice de distance et l’arbre.
On pourra observer les regroupements des espèces les plus proches.
Pour simplifier l’arbre, cliquer sur les boutons contenant les noms des espèces dans le tableau de façon à ne faire apparaître que la tortue, le cheval, le chimpanzé et l’homme.
Cliquer sur le bouton
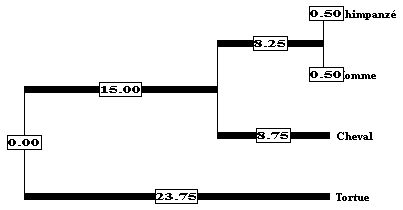
![]() de façon à faire apparaître les distances génétiques entre les espèces. Ces distances sont calculées à partir du nombre d’acides aminés différents entre les différentes molécules. Il y a une différence entre le chimpanzé et l’homme soit 0,5 sur chaque branche.
de façon à faire apparaître les distances génétiques entre les espèces. Ces distances sont calculées à partir du nombre d’acides aminés différents entre les différentes molécules. Il y a une différence entre le chimpanzé et l’homme soit 0,5 sur chaque branche.
Voici l’arbre obtenu avec le fichier myoglobine.pir :
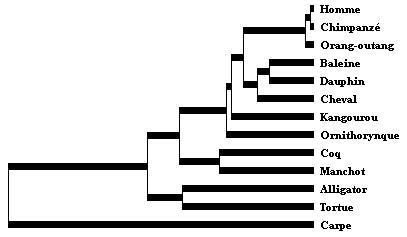
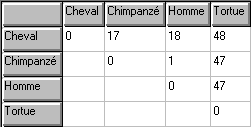
Après simplification, on a la matrice de distances et l’arbre suivants :
 |
 |
On constate qu’il y a, par exemple, 48 acides aminés différents sur 153 entre les myoglobines de tortue (ici la tortue caouanne Caretta caretta) et de cheval. Comment ces deux molécules peuvent-elles encore avoir la même fonction avec environ 1/3 des acides aminés différents ?
On peut visualiser ces différences dans RasTop, pour cela, il faut relever toutes leurs positions.
Ceci peut se faire soit dans Phylogène avec le fichier précédent, soit dans Anagène, après comparaison simple grâce au fichier myoglo.edi.
Télécharger les myoglobines de tortue et de cheval sous forme d’un fichier exe. Enregistrer ce fichier dans le répertoire voulu, faire un double clic, il se décompactera automatiquement en donnant deux fichiers pdb à ouvrir dans RasTop.
| Myoglobine de tortue | Myoglobine de cheval |
|
Les positions des acides aminés différents sont les suivantes :
5, 8-9, 12-13, 15, 19, 21-23, 29, 34-35, 40, 42, 44, 48, 50, 52-55, 57, 60-61, 67, 74, 78, 80-81, 84, 91, 101, 108-110, 112-113, 115-117, 121, 127, 129, 132, 142, 144, 149.
Les acides aminés situés à moins de 5 angströms de l’hème occupent les positions :
32, 39, 42, 43, 45, 64, 67, 68, 71, 72, 89, 92, 93, 97, 99, 103, 104, 107, 111, 138.
On peut constater que deux acides aminés différents seulement sont proches de l’hème mais pas dans la zone de fixation du dioxygène.
| Tortue | Arg 42 | Thr 67 |
| Cheval | Lys 42 | Val 67 |
Le script 1 est un fichier autodécompactable donnant des fichiers rsm à ouvrir comme scripts dans RasTop permettant de mettre en évidence automatiquement les acides aminés différents (magenta) et identiques (bleus) dans les myoglobines de tortue et de cheval.
Le script 2 est un fichier autodécompactable donnant des fichiers rsm à ouvrir comme scripts dans RasTop permettant d’ajouter au précédent les acides aminés proches de l’hème qu’ils soient différents (verts) ou identiques (jaunes) dans les myoglobines de tortue et de cheval.